Et team av bioingeniører har lykkes med å bruke kunstig intelligens til å gjenopplive molekyler fra vår fortid.
Denne spennende studien benyttet maskinlæring (ML) til å analysere proteindata fra moderne mennesker (Homo sapiens) samt fra våre utdødde slektninger, neandertalerne (Homo neanderthalensis) og denisovanere, en utdødd art eller underart av mennesker fra Asia.
Den studie oppdaget molekyler med potensial til å bekjempe sykdomsfremkallende bakterier. Disse teknikkene, kalt "de-extinction", kan potensielt danne grunnlaget for å finne nye antibiotika.
"Vi er motivert av tanken på å bringe tilbake molekyler fra fortiden for å løse problemer vi har i dag", sier Cesar de la Fuente, medforfatter av studien og bioingeniør ved University of Pennsylvania i Philadelphia, fortalte naturen.
Hvis dette får deg til å tenke på en viss dinosaurbasert filmserie - Det var nettopp det som inspirerte forskerteamet.
De la Fuente sa: "Vi begynte faktisk å tenke på Jurassic Parksier han. I stedet for å vekke dinosaurene til live, slik forskerne gjorde i filmen fra 1993, kom teamet opp med en mer gjennomførbar idé: "Hvorfor ikke vekke molekyler til live?"
Det er et enormt press på forskerne for å finne nye antibiotika, og de siste 30 årene har det blitt oppdaget få effektive legemidler.
Samtidig øker forekomsten av antibiotikaresistente bakterier, så nye behandlingsmetoder er kjærkomne. AI gir mange muligheter for å utforske disse nye behandlingene, med en rekke studier som utnytter teknologien til å avdekke skjulte og ukjente klinisk relevante molekyler.
Mange organismer danner korte proteinunderenheter, såkalte peptider, med antimikrobielle egenskaper. Flere antimikrobielle peptider, hovedsakelig fra bakterier, brukes til å behandle infeksjoner.
Proteiner fra utdødde arter - inkludert mennesker - kan være en verdifull, men underutforsket ressurs for utvikling av nye antibiotika.
Forskerne trente opp en ML-algoritme for å identifisere steder på humane proteiner der de er delt inn i peptider. De brukte denne algoritmen på offentlig tilgjengelige proteinsekvenser fra H. sapiens, H. neanderthalensisog denisovaner, og identifiserer nye peptider basert på egenskapene til kjente antimikrobielle peptider.
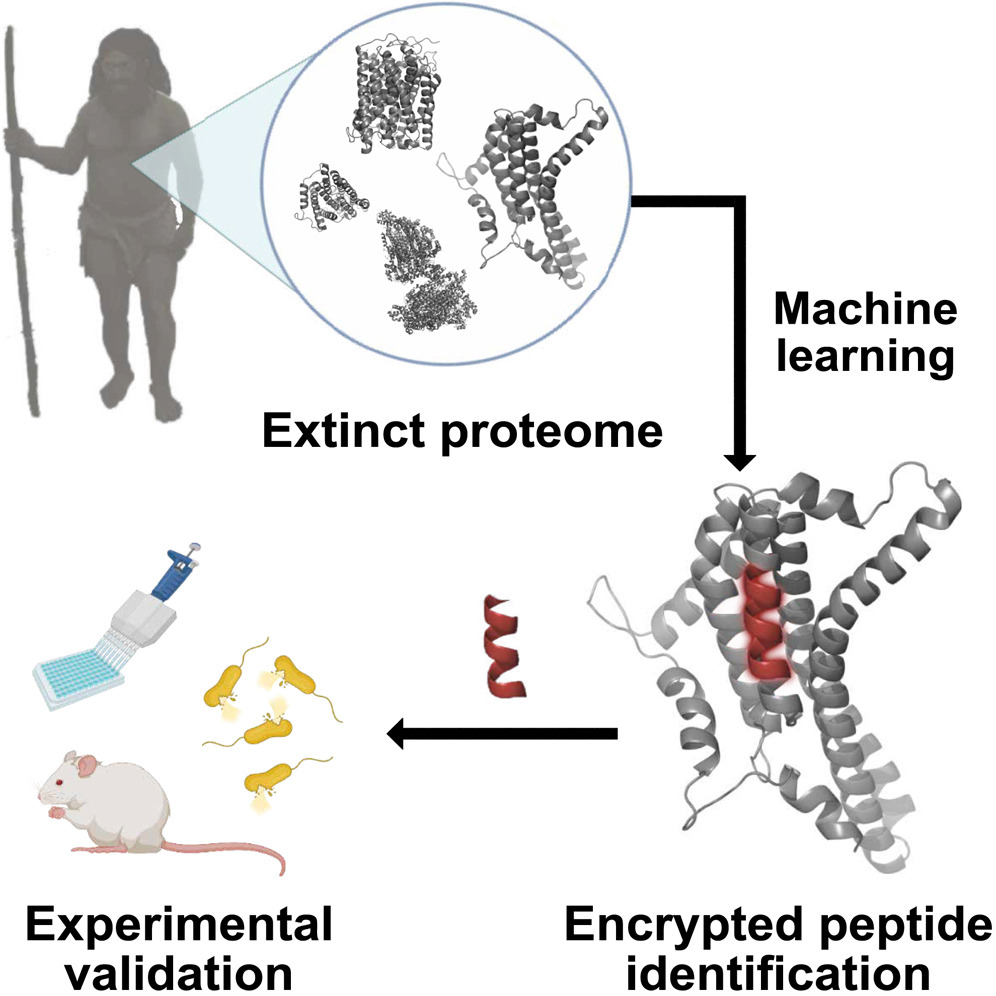
Det tar bare noen få uker å identifisere og teste potensielle legemiddelkandidater ved hjelp av kunstig intelligens, sammenlignet med den tradisjonelle prosessen som tar fra tre til seks år å oppdage et nytt antibiotikum.
Teamet testet en rekke peptider i laboratoriet for å undersøke deres evne til å eliminere bakterier. De valgte ut 6 potente peptider - 4 fra H. sapiens, 1 fra H. neanderthalensisog 1 fra denisovanere. Peptidene ble testet på mus infisert med Acinetobacter baumannii.
Selv om ingen av peptidene drepte bakteriene fullstendig, klarte de alle å hemme veksten av A. baumannii i lårmuskelvev. 5 molekyler drepte bakterier i hudabscesser, selv om det var nødvendig med høye doser.
"Selv om algoritmen vi brukte, ikke ga noen fantastiske molekyler, tror jeg konseptet og rammeverket representerer en helt ny måte å tenke på når det gjelder oppdagelse av legemidler", sier de la Fuente.
Euan Ashley, ekspert på genomikk og presisjonshelse ved Stanford University, kommenterte: "De la Fuente og kollegene hans overbeviste meg om at det å dykke ned i det arkaiske menneskelige genomet var en interessant og potensielt nyttig tilnærming."
Hvordan studien fungerte: en oversikt
- Forskerne satte seg fore å gjenopplive (eller "utdø") bioaktive molekyler som ikke lenger produseres av levende organismer. Disse molekylene kan potensielt brukes til å finne nye legemidler.
- De brukte en maskinlæringsmodell (ML) som kalles panCleave. Dette er en "random forest"-modell, en type AI-algoritme som forutsier utfall basert på mange inndata. PanCleave-modellen forutsa hvor proteiner ville bli delt opp i mindre deler, såkalte peptider. Disse peptidene har noen ganger antimikrobielle egenskaper.
- Resultatet: Forskerne fant flere peptider (både fra moderne mennesker og utdødde slektninger) som viste seg å ha potensial som antimikrobielle midler når de ble testet i laboratoriet.
- Testene omfattet motstand mot å bli brutt ned av andre proteiner og evnen til å trenge gjennom cellemembraner. Noen av peptidene bekjempet effektivt en vanlig bakterieinfeksjon i hudabscesser hos mus.
- Maskinlæringsmetoder kan identifisere lovende peptider for utvikling av nye antibiotika. I tillegg foreslås "molekylær de-extinction" (gjenoppliving av molekyler fra utdødde organismer) som et nytt rammeverk for legemiddeloppdagelse, spesielt for å finne nye antibiotika.
AIs potensial for å oppdage nye legemidler i stor skala viser seg å være enormt, og ikke bare antibiotika, men anti-aldringsmedisiner og andre forbindelser også.





