Uma equipa de bioengenheiros utilizou com sucesso a IA para ressuscitar moléculas do nosso passado antigo.
Este estudo intrigante utilizou a aprendizagem automática (ML) para analisar dados de proteínas de humanos modernos (Homo sapiens), bem como dos nossos parentes extintos, os Neandertais (Homo neanderthalensis) e os Denisovans, uma espécie ou subespécie extinta de humanos da Ásia.
O estudo descobriram moléculas com potencial para combater bactérias causadoras de doenças. Estas técnicas, designadas por "de-extinção", poderão servir de base para a localização de novos antibióticos.
"Estamos motivados pela noção de trazer de volta moléculas do passado para resolver problemas que temos atualmente", Cesar de la Fuente, coautor do estudo e bioengenheiro da Universidade da Pensilvânia, em Filadélfia, disse à Nature.
Se isto o faz pensar numa certa franquia de filmes baseados em dinossauros - foi precisamente isso que inspirou a equipa de investigação.
De la Fuente disse: "Começámos a pensar em Parque Jurássico", diz ele. Em vez de trazer os dinossauros de volta à vida, como os cientistas fizeram no filme de 1993, a equipa teve uma ideia mais viável: "Porque não trazer de volta as moléculas?
A pressão exercida sobre os investigadores para encontrarem novos antibióticos é enorme, uma vez que nos últimos 30 anos foram descobertos poucos medicamentos eficazes.
Entretanto, a prevalência de bactérias resistentes aos antibióticos está a aumentar, pelo que são bem-vindos novos tratamentos. A IA fornece numerosas vias para explorar esses novos tratamentos, com uma série de estudos que tiram partido da tecnologia para descobrir moléculas ocultas e desconhecidas clinicamente relevantes.
Muitos organismos geram subunidades proteicas curtas, conhecidas como péptidos, com propriedades antimicrobianas. Vários péptidos antimicrobianos, predominantemente provenientes de bactérias, são utilizados para tratar infecções.
As proteínas de espécies extintas - incluindo os seres humanos - podem servir como um recurso valioso, mas pouco explorado, para o desenvolvimento de novos antibióticos.
Os investigadores treinaram um algoritmo de aprendizagem de máquina para identificar os locais nas proteínas humanas onde estas são divididas em péptidos. Aplicaram este algoritmo a sequências de proteínas publicamente disponíveis de H. sapiens, H. neanderthalensise Denisovans, identificando novos péptidos com base nas características de péptidos antimicrobianos conhecidos.
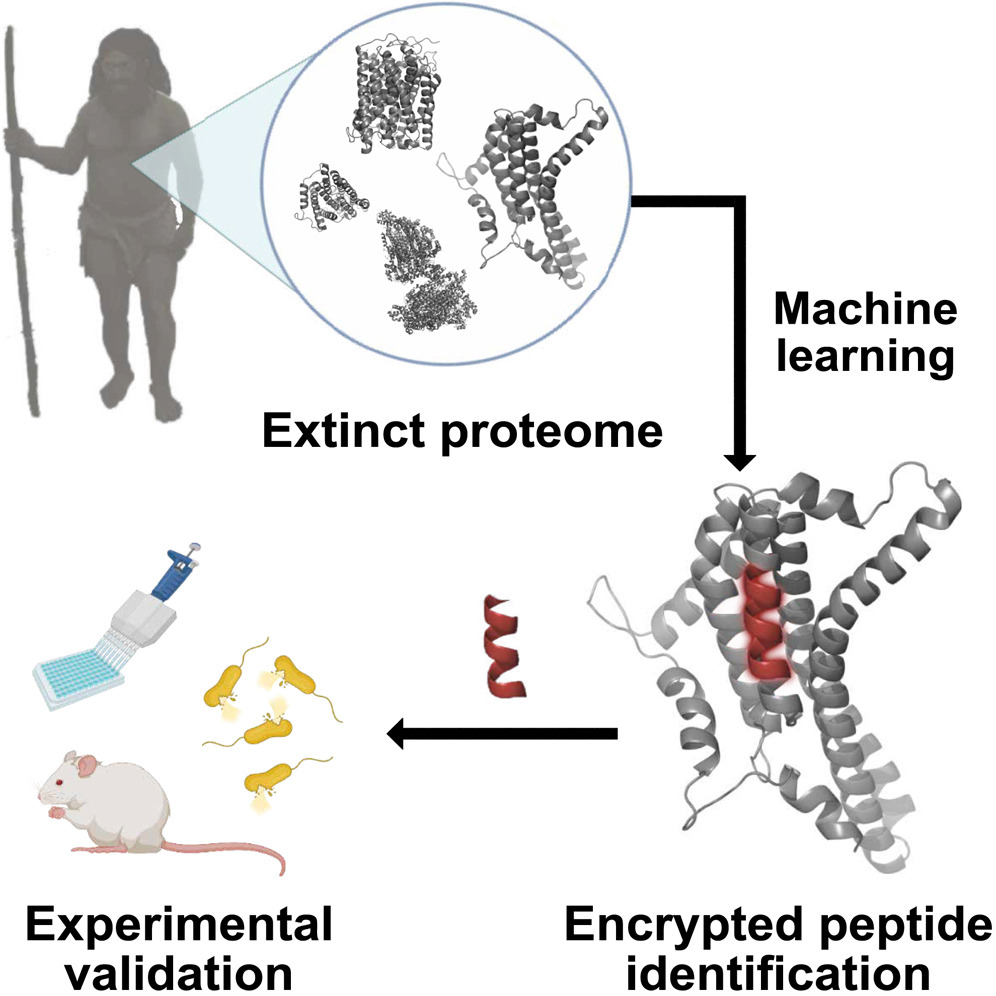
São necessárias apenas algumas semanas para identificar e testar potenciais candidatos a medicamentos utilizando a IA, em comparação com o processo tradicional que demora entre 3 a 6 meses para descobrir um novo antibiótico.
A equipa testou uma variedade de péptidos em laboratório para explorar a sua capacidade de eliminar bactérias. Seleccionaram 6 péptidos potentes - 4 de H. sapiens, 1 de H. neanderthalensise 1 de Denisovans. Os péptidos foram testados em ratinhos infectados com Acinetobacter baumannii.
Embora nenhum dos peptídeos tenha matado completamente as bactérias, todos eles conseguiram inibir o crescimento de A. baumannii no tecido muscular da coxa. 5 moléculas mataram bactérias em abcessos cutâneos, embora fossem necessárias doses elevadas.
"Embora o algoritmo que utilizámos não tenha produzido moléculas fantásticas, penso que o conceito e a estrutura representam uma via totalmente nova para pensar na descoberta de medicamentos", afirma de la Fuente.
Euan Ashley, especialista em genómica e saúde de precisão da Universidade de Stanford, comentou: "De la Fuente e os seus colegas persuadiram-me de que mergulhar no genoma humano arcaico era uma abordagem interessante e potencialmente útil".
Como funcionou o estudo: resumo
- Os investigadores propuseram-se ressuscitar (ou "de-extinção") moléculas bioactivas que já não são produzidas por organismos vivos. Estas moléculas poderiam ser utilizadas para a descoberta de medicamentos.
- Utilizaram um modelo de aprendizagem automática (ML) conhecido como panCleave. Trata-se de um modelo de "floresta aleatória", um tipo de algoritmo de IA que prevê resultados com base em muitos dados. O modelo panCleave previu onde as proteínas seriam divididas em partes mais pequenas, conhecidas como péptidos. Estes péptidos têm, por vezes, propriedades antimicrobianas.
- O resultado: Os investigadores encontraram vários péptidos (tanto de humanos modernos como de parentes humanos extintos) que mostraram potencial como agentes antimicrobianos quando testados em laboratório.
- Os testes incluíram a resistência a ser quebrada por outras proteínas e a capacidade de permear as membranas celulares. Alguns dos péptidos combateram eficazmente uma infeção bacteriana comum em abcessos cutâneos de ratinhos.
- Os métodos de aprendizagem automática podem identificar péptidos promissores para o desenvolvimento de novos antibióticos. Além disso, a "de-extinção molecular" (ressuscitar moléculas de organismos extintos) é proposta como um novo quadro para a descoberta de medicamentos, especificamente para encontrar novos antibióticos.
O potencial da IA para a descoberta de novos medicamentos à escala está a revelar-se imenso, e não apenas para os antibióticos, mas medicamentos anti-envelhecimento e outros compostos também.





