Forskere ved University of Cambridge har udnyttet AI i kampen mod antibiotikaresistens.
Forskerteamet, ledet af professor Stephen Baker, skabte et maskinlæringsværktøj, der kun bruger mikroskopibilleder til at skelne mellem bakterier, der er resistente over for ciprofloxacin (et almindeligt antibiotikum), og dem, der er modtagelige over for det.
Det kan dramatisk reducere den tid, det tager at diagnosticere antibiotikaresistens, og potentielt ændre den måde, vi behandler farlige infektioner som tyfus på.
Den undersøgelsesom blev offentliggjort i Nature Communications, fokuserede på Salmonella Typhimurium, en bakterie, der forårsager alvorlig mave-tarm-sygdom og kan føre til livstruende invasiv sygdom.
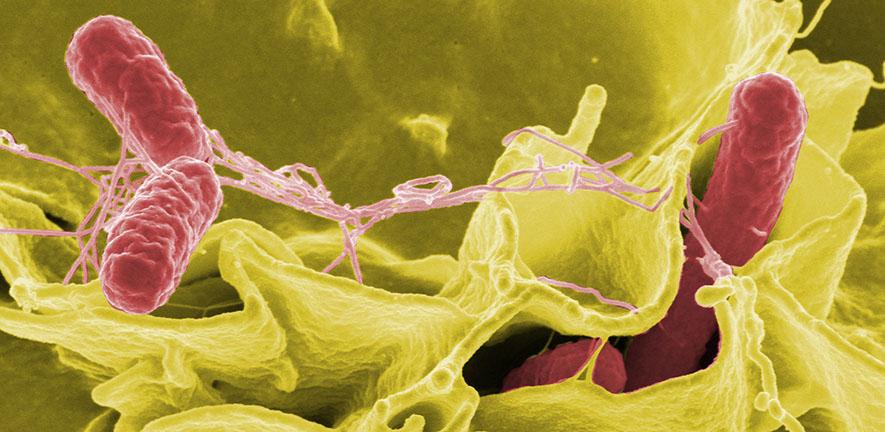
Dr. Tuan-Anh Tran, en af projektets nøgleforskere, forklarede tilgangen i en blogindlæg: "Det smukke ved maskinlæringsmodellen er, at den kan identificere resistente bakterier ud fra nogle få subtile træk på mikroskopibilleder, som det menneskelige øje ikke kan opdage."
Forskningsprocessen omfattede flere vigtige trin:
- Forberedelse af bakterieprøver: Holdet dyrkede S. Typhimurium-prøver i flydende næringsmedier, hvor nogle blev udsat for forskellige koncentrationer af ciprofloxacin og andre ikke.
- Billeddannelse med højt indhold: Ved hjælp af et sofistikeret mikroskop tog forskerne detaljerede billeder af bakterierne på flere tidspunkter.
- Billedanalyse: Specialiseret software udtrak 65 forskellige funktioner fra hver bakteriecelle, herunder form, størrelse og interaktion med fluorescerende farvestoffer.
- Udvikling af maskinlæringsmodeller: Forskerne førte disse data ind i forskellige maskinlæringsalgoritmer og trænede dem i at genkende mønstre, der er forbundet med antibiotikaresistens.
- Udvælgelse af funktioner: Holdet identificerede de mest afgørende træk til at skelne mellem resistente og modtagelige bakterier.
Resultaterne af denne proces var imponerende. AI-systemet identificerede antibiotikaresistente bakterier korrekt i ca. 87% af tilfældene.
Måske er det mest bemærkelsesværdige, at forskerne fandt ud af, at resistente og modtagelige bakterier havde forskellige visuelle mønstre, som AI'en kunne registrere, selv når de ikke havde været udsat for antibiotika.
Det tyder på, at antibiotikaresistens ændrer bakteriernes udseende på måder, der er for subtile til, at mennesker kan se dem, men som kunstig intelligens kan opdage.
De nuværende metoder kræver typisk flere dages bakteriekultur og test mod forskellige antimikrobielle stoffer. I modsætning hertil kan den nye AI-baserede metode potentielt give resultater inden for få timer.
Hurtigere diagnose gør det muligt for lægerne at ordinere de mest effektive antibiotika hurtigere, hvilket potentielt kan forbedre patientresultaterne og reducere spredningen af resistente bakterier.
Fremover vil forskerholdet udvide deres tilgang til mere komplekse kliniske prøver som blod eller urin og teste dem på andre typer bakterier og antibiotika. De arbejder også på at gøre teknologien mere tilgængelig for hospitaler og klinikker verden over.
Som professor Baker forklarer: "Det, der ville være virkelig vigtigt, især i en klinisk sammenhæng, ville være at kunne tage en kompleks prøve - for eksempel blod, urin eller sputum - og identificere modtagelighed og resistens direkte ud fra den."
"Det er et meget mere kompliceret problem, og det er slet ikke blevet løst, ikke engang inden for klinisk diagnostik på et hospital. Hvis vi kunne finde en måde at gøre det på, kunne vi reducere den tid, det tager at identificere lægemiddelresistens, og til en meget lavere pris. Det kunne virkelig være transformerende."
Dr. Sushmita Sridhar opsummerede virkningerne og sagde: "I betragtning af at denne tilgang bruger billeddannelse med enkeltcelleopløsning, er det endnu ikke en løsning, der let kan implementeres overalt. Men det er virkelig lovende, at vi ved at indfange blot nogle få parametre om bakteriernes form og struktur kan give os nok information til relativt nemt at forudsige lægemiddelresistens."
Da antibiotikaresistens fortsat udgør en eskalerende global sundhedstrussel, giver innovative tilgange som denne AI-drevne billeddannelsesteknik nyt håb.
Dette er en del af en bredere tendens med AI-drevne innovationer inden for antibiotikaforskning. På MIT har forskere brugt deep learning-modeller til at opdage en Helt ny klasse af antibiotika.
På samme måde meddelte et andet hold forskere i maj sidste år, at de havde brugt AI til at identificere et nyt antibiotikum effektiv mod resistente bakterier.
AI muliggør hurtigere og mere præcis identifikation af lægemiddelresistente infektioner, hvilket baner vejen for mere effektive behandlinger og bedre patientresultater.
De næste par år bliver afgørende, da teamet arbejder på at omsætte deres laboratoriesucces til kliniske anvendelser i den virkelige verden.





