Ett team av bioingenjörer har framgångsrikt använt AI för att återuppliva molekyler från vårt forntida förflutna.
Denna spännande studie använde maskininlärning (ML) för att analysera proteindata från moderna människor (Homo sapiens) samt från våra utdöda släktingar, neandertalarna (Homo neanderthalensis) och Denisovaner, en utdöd art eller underart av människor från Asien.
Den studie upptäckt molekyler med potential att bekämpa sjukdomsframkallande bakterier. Dessa tekniker, som kallas "de-extinction", skulle potentiellt kunna ligga till grund för att hitta nya antibiotika.
"Vi motiveras av tanken på att föra tillbaka molekyler från det förflutna för att lösa problem som vi har idag", säger Cesar de la Fuente, medförfattare till studien och bioingenjör vid University of Pennsylvania i Philadelphia, berättade för Nature.
Om detta får dig att tänka på en viss dinosauriebaserad filmserie - Det var just det som inspirerade forskargruppen.
De la Fuente sa: "Vi började faktiskt fundera på Jurassic Park", säger han. I stället för att återuppliva dinosaurier, som forskarna gjorde i filmen från 1993, kom teamet på en mer genomförbar idé: "Varför inte återuppliva molekyler?"
Det finns ett enormt tryck på forskarna att hitta nya antibiotika, och få effektiva läkemedel har upptäckts under de senaste 30 åren.
Samtidigt ökar förekomsten av antibiotikaresistenta bakterier, vilket gör att nya behandlingsmetoder är välkomna. AI tillhandahåller många möjligheter för att utforska dessa nya behandlingar, med en rad studier som utnyttjar tekniken för att avslöja dolda och okända kliniskt relevanta molekyler.
Många organismer genererar korta proteinsubenheter, så kallade peptider, med antimikrobiella egenskaper. Flera antimikrobiella peptider, som främst kommer från bakterier, används för att behandla infektioner.
Proteiner från utdöda arter - inklusive människan - kan utgöra en värdefull men ännu outforskad resurs för att utveckla nya antibiotika.
Forskarna tränade en ML-algoritm för att identifiera platser på mänskliga proteiner där de skärs upp i peptider. De tillämpade denna algoritm på offentligt tillgängliga proteinsekvenser från H. sapiens, H. neanderthalensisoch Denisovans, och identifierade nya peptider baserat på egenskaperna hos kända antimikrobiella peptider.
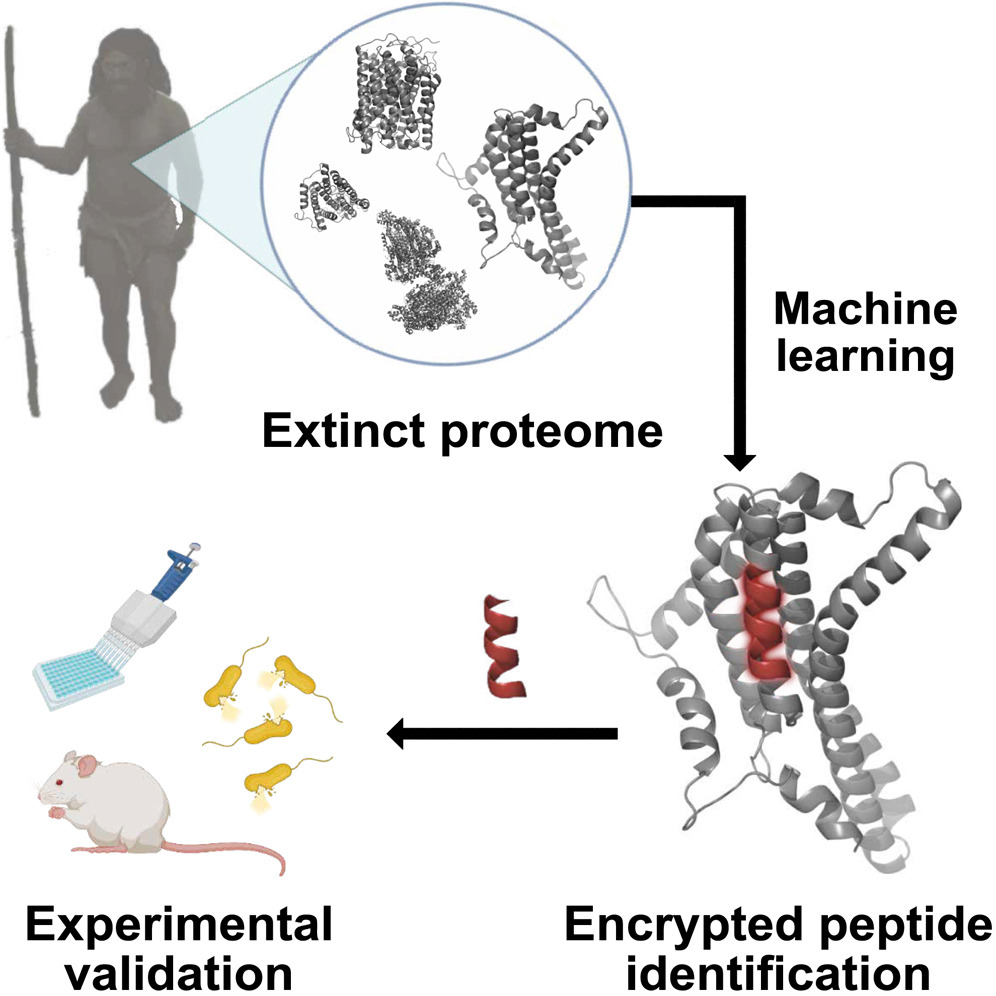
Det tar bara några veckor att identifiera och testa potentiella läkemedelskandidater med hjälp av AI, jämfört med den traditionella processen som tar mellan 3 och 6 månader för att upptäcka ett nytt antibiotikum.
Teamet testade en rad olika peptider i laboratoriemiljö för att undersöka deras förmåga att eliminera bakterier. De valde ut 6 potenta peptider - 4 från H. sapiens, 1 från H. neanderthalensisoch 1 från Denisovans. Peptiderna testades på möss som var infekterade med Acinetobacter baumannii.
Även om ingen av peptiderna helt dödade bakterierna lyckades de alla hämma tillväxten av A. baumannii i muskelvävnad i låret. 5 molekyler dödade bakterier i hudabscesser, även om det krävdes höga doser.
"Även om den algoritm som vi använde inte gav några fantastiska molekyler, tror jag att konceptet och ramverket utgör en helt ny väg för att tänka på läkemedelsupptäckt", säger de la Fuente.
Euan Ashley, expert på genomik och precisionshälsa vid Stanford University, kommenterade: "De la Fuente och hans kollegor övertygade mig om att det var en intressant och potentiellt användbar metod att dyka ner i det arkaiska mänskliga genomet."
Hur studien fungerade: en sammanfattning
- Forskarna ville återuppliva (eller "de-extinct") bioaktiva molekyler som inte längre produceras av levande organismer. Dessa molekyler skulle potentiellt kunna användas för läkemedelsupptäckt.
- De använde en maskininlärningsmodell (ML) som kallas panCleave. Detta är en "random forest"-modell, en typ av AI-algoritm som förutspår resultat baserat på många indata. PanCleave-modellen förutspådde var proteiner skulle delas upp i mindre delar, så kallade peptider. Dessa peptider har ibland antimikrobiella egenskaper.
- Resultatet: Forskarna hittade flera peptider (både från moderna människor och utdöda släktingar till människan) som visade potential som antimikrobiella medel när de testades i labbet.
- Bland annat testades motståndskraften mot att brytas ned av andra proteiner och förmågan att tränga igenom cellmembran. Några av peptiderna bekämpade effektivt en vanlig bakterieinfektion i hudabscesser hos möss.
- Maskininlärningsmetoder kan identifiera lovande peptider för utveckling av nya antibiotika. Dessutom föreslås "molekylär de-extinction" (återuppväckande av molekyler från utdöda organismer) som ett nytt ramverk för läkemedelsupptäckt, särskilt för att hitta nya antibiotika.
AI:s potential för att upptäcka nya läkemedel i stor skala visar sig vara enorm, och inte bara antibiotika utan läkemedel mot åldrande och andra föreningar också.





