Un equipo de bioingenieros ha utilizado con éxito la IA para resucitar moléculas de nuestro antiguo pasado.
Este interesante estudio utilizó el aprendizaje automático (ML) para analizar datos sobre proteínas de humanos modernos (Homo sapiens), así como de nuestros parientes extintos, los neandertales (Homo neanderthalensis) y los denisovanos, una especie o subespecie humana extinguida de Asia.
En estudiar han descubierto moléculas con potencial para combatir bacterias patógenas. Estas técnicas, denominadas "de-extinción", podrían servir de base para localizar nuevos antibióticos.
"Nos motiva la idea de recuperar moléculas del pasado para resolver problemas actuales", César de la Fuente, coautor del estudio y bioingeniero de la Universidad de Pensilvania en Filadelfia, dijo a Nature.
Si esto le hace pensar en cierta franquicia cinematográfica basada en dinosaurios - Eso es precisamente lo que inspiró al equipo de investigación.
De la Fuente dijo: "En realidad empezamos a pensar en Parque Jurásicodice. En lugar de resucitar dinosaurios, como hicieron los científicos en la película de 1993, al equipo se le ocurrió una idea más factible: "¿Por qué no resucitar moléculas?".
Existe una inmensa presión sobre los investigadores para localizar nuevos antibióticos, ya que en los últimos 30 años se han descubierto pocos fármacos eficaces.
Mientras tanto, la prevalencia de bacterias resistentes a los antibióticos va en aumento, por lo que los tratamientos novedosos son bienvenidos. La IA proporciona numerosas vías para explorar esos tratamientos novedosos, con una serie de estudios que aprovechan la tecnología para descubrir moléculas ocultas y desconocidas clínicamente relevantes.
Muchos organismos generan subunidades proteicas cortas conocidas como péptidos con propiedades antimicrobianas. Varios péptidos antimicrobianos, procedentes sobre todo de bacterias, se utilizan para tratar infecciones.
Las proteínas de especies extinguidas -incluida la humana- podrían constituir un recurso valioso, aunque poco explorado, para desarrollar nuevos antibióticos.
Los investigadores entrenaron un algoritmo de ML para identificar los lugares de las proteínas humanas donde se cortan en péptidos. Aplicaron este algoritmo a secuencias de proteínas disponibles públicamente de H. sapiens, H. neanderthalensisy Denisovanos, identificando nuevos péptidos basados en las características de péptidos antimicrobianos conocidos.
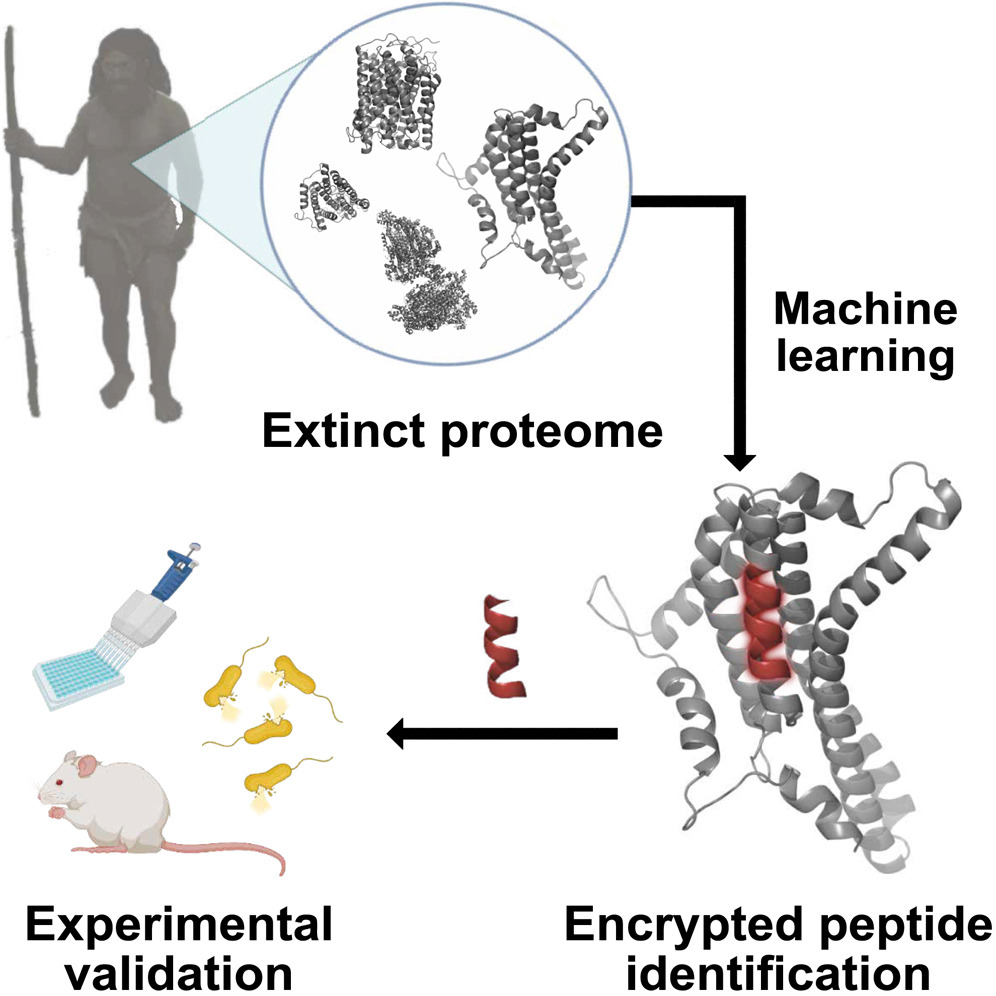
Sólo se tarda unas semanas en identificar y probar posibles candidatos a fármacos mediante la IA, frente al proceso tradicional que tarda entre 3 y 6 6 en descubrir un nuevo antibiótico.
El equipo probó diversos péptidos en un laboratorio para explorar su capacidad de eliminar bacterias. Seleccionaron 6 potentes péptidos - 4 de H. sapiens1 de H. neanderthalensisy 1 de denisovanos. Los péptidos se probaron en ratones infectados con Acinetobacter baumannii.
Aunque ninguno de los péptidos mató por completo a las bacterias, todos consiguieron inhibir el crecimiento de A. baumannii en el tejido muscular del muslo. 5 moléculas mataron bacterias en abscesos cutáneos, aunque se necesitaron dosis elevadas.
"Aunque el algoritmo que utilizamos no produjo moléculas asombrosas, creo que el concepto y el marco representan una vía completamente nueva para pensar en el descubrimiento de fármacos", afirma de la Fuente.
Euan Ashley, experto en genómica y salud de precisión de la Universidad de Stanford, comentó: "De la Fuente y sus colegas me convencieron de que bucear en el genoma humano arcaico era un enfoque interesante y potencialmente útil."
Cómo ha funcionado el estudio: desglose
- Los investigadores se propusieron resucitar (o "eliminar") moléculas bioactivas que ya no producen los organismos vivos. Estas moléculas podrían utilizarse para descubrir fármacos.
- Utilizaron un modelo de aprendizaje automático conocido como panCleave. Se trata de un modelo de "bosque aleatorio", un tipo de algoritmo de inteligencia artificial que predice resultados basándose en muchos datos. El modelo panCleave predijo dónde se dividirían las proteínas en partes más pequeñas conocidas como péptidos. Estos péptidos tienen a veces propiedades antimicrobianas.
- El resultado: Los investigadores encontraron varios péptidos (tanto de humanos modernos como de parientes humanos extintos) que mostraron potencial como agentes antimicrobianos cuando se probaron en el laboratorio.
- Las pruebas incluyeron la resistencia a la rotura por otras proteínas y la capacidad de permeabilizar las membranas celulares. Algunos de los péptidos combatieron eficazmente una infección bacteriana común en abscesos cutáneos de ratones.
- Los métodos de aprendizaje automático pueden identificar péptidos prometedores para el desarrollo de nuevos antibióticos. Además, se propone la "desextinción molecular" (resucitar moléculas de organismos extintos) como nuevo marco para el descubrimiento de fármacos, concretamente para hallar nuevos antibióticos.
El potencial de la IA para descubrir nuevos fármacos a escala está resultando inmenso, y no sólo antibióticos, sino medicamentos antienvejecimiento y otros compuestos también.





